In Science ist ein Bericht zu einer neuen Arbeit veröffentlicht worden, in der ein internationales Forscherteam beschreibt, dass es 18 neue HSP-Gene gefunden hat. Diese Menge war nur möglich, weil ein Verfahren ‒nämlich die Exom-Sequenzierung‒ angewandt wurde. Bei der Exom-Sequenzierung wird die Gesamtmenge aller Gene untersucht, die die Information für Proteine in sich tragen; es wird hier nicht mehr nur in zuvor bestimmten Bereichen gesucht. Allein die Menge der neu gefundenen HSP-Gene zeigt, dass die Aussage von Herrn Prof. Züchner (Miami), dass es bei der HSP etwa 200 unterschiedliche Gene geben wird, wirklich zutreffend sein wird.
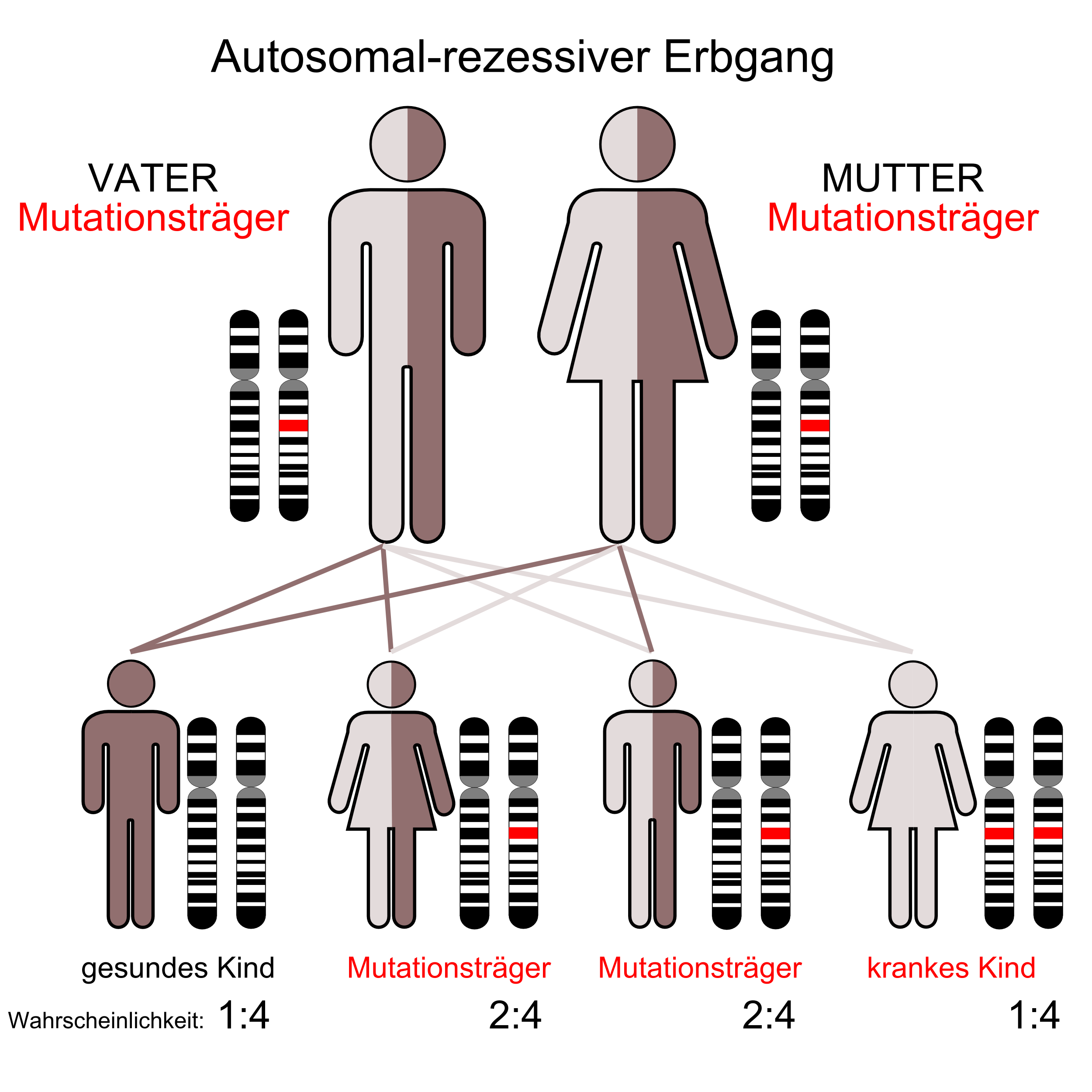
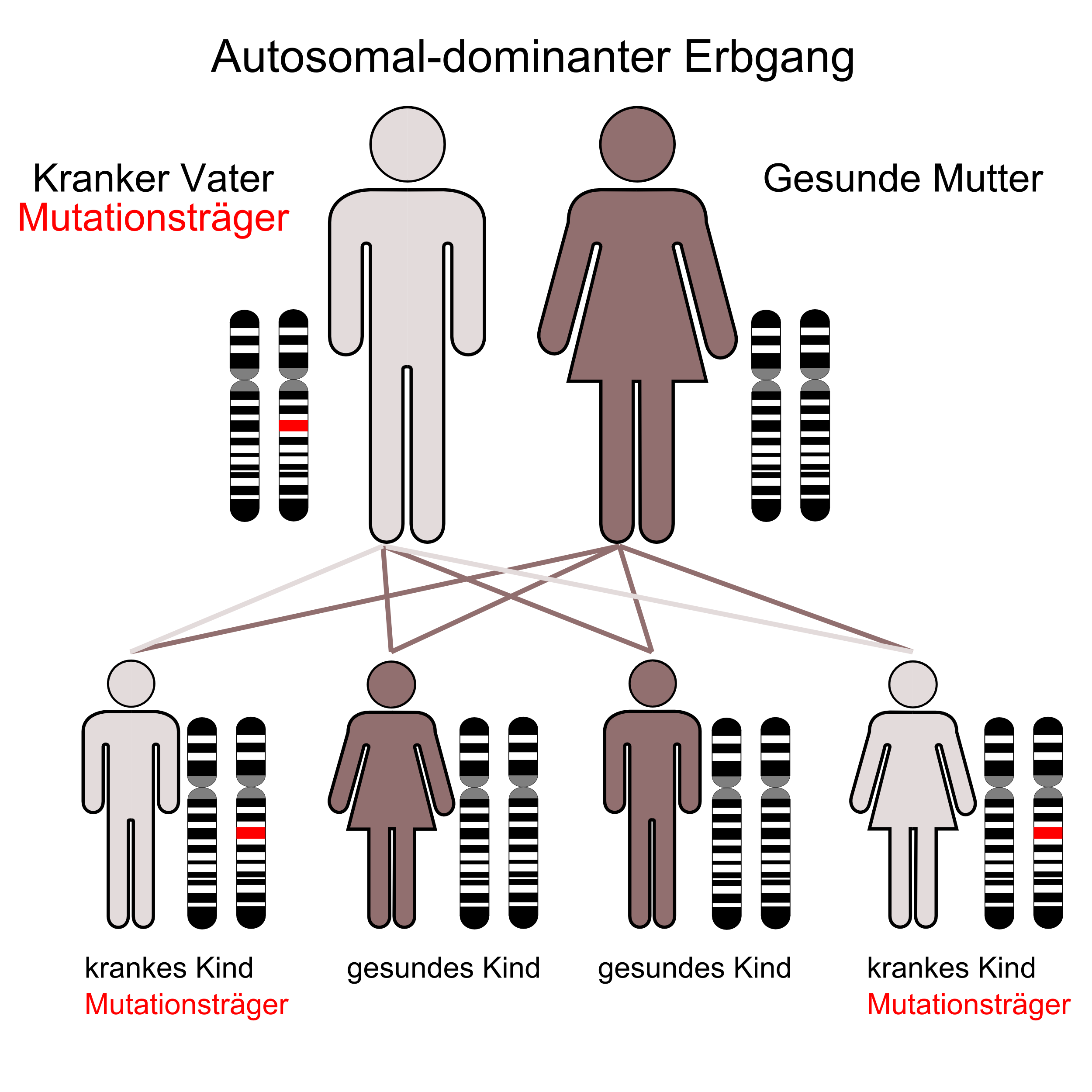
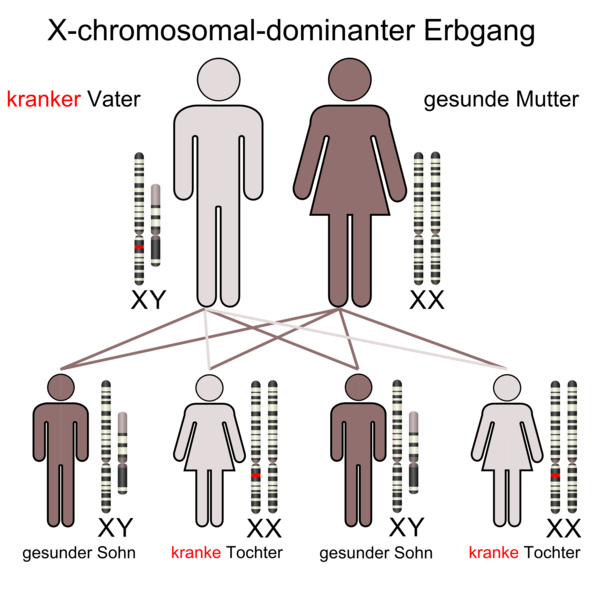
Die gefundenen 18 neuen HSP-Gene sind alle rezessive Gene. Auch dadurch wird erneut unterlegt, dass die Menge der rezessiven HSP-Gene wesentlich höher sein könnte, als die Menge der dominanten HSP-Gene. Dem Grunde nach ist das aber seit einigen Jahren bereits immer wieder angesprochen worden; man muss diese vielen Gene nur finden. Und ganz wesentlich ist es natürlich, dass jedes Gen neue Erkenntnisse zur HSP bringt, die helfen werden, therapeutische Lösungen zu finden. Allein deshalb ist jedes neu gefundene HSP-Gen ‒gleichgültig ob es rezessiv, dominant oder X-chromosomal ist‒ auch eine Hoffnung für die HSP-Erkrankten.
Y-chromosomaler Erbgang
Es sind beim Menschen, im Gegensatz zu den X-chromosomalen Erbgängen, bei dem Y-chromosomaler Erbgang keine Erbkrankheiten bekannt. Daher ist hier auch kein erklärendes Bild eingestellt.
Unten der Text der Zusammenfassung im Original und in automatischer deutscher Übersetzung. Zusätzlich die Links zu Zeitungsartikeln (bisher nur in Englisch), die dieses Thema aufgegriffen haben
——————————————-

Science 31 January 2014
Exome Sequencing Links Corticospinal Motor Neuron Disease to Common Neurodegenerative Disorders
Hereditary spastic paraplegias (HSPs) are neurodegenerative motor neuron diseases characterized by progressive age-dependent loss of corticospinal motor tract function. Although the genetic basis is partly understood, only a fraction of cases can receive a genetic diagnosis, and a global view of HSP is lacking. By using whole-exome sequencing in combination with network analysis, we identified 18 previously unknown putative HSP genes and validated nearly all of these genes functionally or genetically. The pathways highlighted by these mutations link HSP to cellular transport, nucleotide metabolism, and synapse and axon development. Network analysis revealed a host of further candidate genes, of which three were mutated in our cohort. Our analysis links HSP to other neurodegenerative disorders and can facilitate gene discovery and mechanistic understanding of disease.
Quelle: http://www.sciencemag.org/content/343/6 … ref-fn-1-1
——————————————————–
Exom Sequencierung stellt Verbindung zwischen corticospinaler Motorneuron-Krankheit zu allgemeinen neurodegenerative Erkrankungen her
Hereditäre Spastische Paraplegien (HSP) sind neurodegenerative Motorneuron-Krankheiten, die durch einen fortschreitenden, altersabhängigen Funktionsverlust im corticospinalen Nervenstrang charakterisiert sind. Obwohl die genetischen Grundlagen teilweise verstanden wird, kann nur in einem Bruchteil der Fälle eine genetische Diagnose erreicht werden, und eine globale Sicht auf HSP fehlt. Durch die Verwendung von vollständigen Exom-Sequenzierungen in Kombination mit einer Netzwerkanalyse identifizierten wir 18 bisher unbekannte, mutmaßliche HSP-Gene und validierten fast alle diese Gene funktionell oder genetisch. Die von diesen Mutationen hervorgerufenen Veränderungen zeigen Zusammenhänge der HSP mit dem zellulären Transport, mit dem Nukleotid-Stoffwechsel und mit der Entwicklung der Synapsen und Axone. Die Netzwerk-Analyse ergab eine Vielzahl weiterer Kandidatengene, von denen drei in unserer Kohorte mutiert waren. Unsere Analyse verbindet HSP mit anderen neurodegenerative Krankheiten und kann die Genanalyse und das mechanistische Verständnis der Krankheit erleichtern.
—————————————————–
Und hier Zeitungsartikel zum Thema
New genetic forms of neurodegeneration discovered
______________________________________________________________________________________
Zu der wichtigen Nachricht bezüglich der 18 neu gefundenen HSP-Gene gibt es auch einen Text in deutscher Sprache. Er stammt von der APA – Austria Presse Agentur eG, Wien. Vermutlich wurde das Thema dort auch deshalb aufgegriffen, weil die Arbeit unter Mitwirkung von Gaia Novarino als Erstautorin entstanden ist, die seit Jahresbeginn Assistant Professor am IST Austria tätig ist.

Entdeckung neuer genetischer Formen von Neurodegeneration
31.01.2014
Klosterneuburg (ISTA) – Science-Publikation von IST Austria Assistant Professor Gaia Novarino und KollegInnen enthüllt Zusammenhänge zwischen seltener Störung und häufigen Erkrankungen wie Alzheimer.
In einer Studie, die am 31. Jänner in Science erscheint, berichtet ein internationales Forscher-Team, geleitet von WissenschaftlerInnen an der University of California San Diego School of Medicine, von einer Verdopplung der Anzahl an bekannten Ursachen für hereditäre spastische Paraplegie (HSP), eine neurodegenerative Erkrankung; die Arbeit entstand unter Mitwirkung von Gaia Novarino als Erstautorin, die seit Jahresbeginn Assistant Professor am IST Austria ist. HSP wird charakterisiert durch eine zunehmende Versteifung und Kontraktion der unteren Gliedmaßen, und wird begleitet von Epilepsie, kognitiven Beeinträchtigungen, Erblindung und, in manchen Fällen, weiteren neurologischen Symptomen.
Über mehrere Jahre hinweg rekrutierte und begleitet das Forscherteam an der UCSD eine Gruppe von mehr als 50 Familien, bei denen autosomal rezessive HSP auftritt: Damit handelt es sich um die bisher größte erforschte Gruppe von Betroffenen. Die WissenschaftlerInnen analysierten 100 PatientInnen dieser Kohorte mithilfe der Exom-Sequenzierung, einer Methode, mit der die wichtigsten Teile des Genoms analysiert werden. In fast 75% der Fälle identifizierten die ForscherInnen eine genetische Mutation. Die Hälfte davon wurde in Genen gefunden, die bisher nicht mit Erkrankungen in Verbindung gebracht worden waren.
„Nachdem wir so viele neue genetische Ursachen von HSP entdeckt hatten, waren wir in der einzigarten Lage, diese Ursachen miteinander in Verbindung zu setzen und ein ‚HSP-ome‘ zu erstellen, also eine Karte, die alle neuen und zuvor beschriebenen Ursachen beinhaltet“, erklärt Seniorautor Joseph G. Gleeson, MD Professor an der UC San Diego und Investigator des Howard Hughes Medical Institute.
Die Erstellung des „HSP-ome“ half dem Team, weitere genetische Mutationen bei ihren PatientInnen zu identifizieren und später zu bestätigen, wodurch maßgebliche biologische Schaltkreise, die HSP zu Grund liegen, bestimmt werden konnten. Darüber hinaus wollten die ForscherInnen verstehen, wie HSP mit anderen Gruppen von Erkrankungen zusammenhängt; sie entdeckten, dass durch das HSP-‚ome‘ häufiger auftretende neurodegenerative Erkrankungen wie Alzheimer und amyotropher Lateralsklerosis mit HSP in Zusammenhang gebracht werden können.
„Das Wissen über biologische Prozesse, die neurodegenerativen Erkrankungen zugrunde liegen, ist Ausschlag gebend, um weitere wissenschaftliche Studien voranzutreiben, die ihrerseits darauf abzielen, die exakten Mechanismen von häufigen neurodegenerativen Erkrankungen aufzudecken, und einen Weg für die Entwicklung wirksamer Therapien aufzuzeigen“ so Joseph G. Gleeson.
„Ich halte diese Studie für sehr wichtig für die Forschungsgemeinschaft, die sich mit neurodegenerativen Erkrankungen befasst“ sagt Erstautorin Gaia Novarino, „aber weiter gefasst illustriert die Studie, wie die Verwendung von Genomik in einer spezifischen PatientInnengruppe, und die darauf aufbauende Erstellung eines ‚Interaktoms‘, unser Wissen um bisher unbekannte Ursachen von Erkrankungen erweitern kann.“
Gaia Novarino kam Anfang 2014 als Assistant Professor an das IST Austria. Novarino studierte Molekularbiologie und erhielt 2006 ihr PhD in Zellbiologie an der Universität La Sapienza in Rom. Novarino forschte vier Jahre als Postdoctoral Fellow am Max-Delbrück-Centrum für Molekulare Medizin in Berlin, bevor sie 2010 an das Labor von Joseph Gleeson an der University of California San Diego wechselte, wo sie sich mit der Identifizierung der genetischen Grundlagen kognitiver Störungen befasste und die jetzt in Science veröffentlichte Forschung durchführte.
Rückfraghinweis:
Oliver Lehmann
Media Relations
Institute of Science and Technology Austria
Phone +43-(0)2243 9000-1006; Fax +43-(0)2243 9000-2000
Mobile +43-(0)676 40 12 562; Skype: ViennaLehmann
Am Campus 1
A-3400 Klosterneuburg
E-Mail: oliver.lehmann@ist.ac.at
Visit our website: http://www.ist.ac.at
Quelle: http://science.apa.at/site/medizin_und_ … 2216728862